Regresión entre series y regresiones con lag.¶
En el cuaderno anterior vimos el el Modelo de Regresión Lineal:
En el modelo:
\(\beta_1\ldots\beta_q\) son los parámetros a ajustar
\(z_{t_i}\) son las funciones de regresión a usar, evaluadas en cada \(t\).
\(w_t\) es ruido blanco (gaussiano) de varianza \(\sigma_w^2\).
Usando el comando ols de statsmodels.formula.api se ajustan modelos lineales como el anterior.
Vimos además algunos de sus usos:
Ajustar funciones de tendencia a series temporales.
Hacer regresión con “factores” que representen fenómenos especiales (ej: trimestres), (Ejercicio 1).
Ajustar componentes periódicas (Ejercicio 2)…más sobre esto en este cuaderno.
Queremos ahora estudiar:
El uso de modelos lineales para hallar relaciones entre series que permitan explicar una a partir de otra.
Cómo ajustar componentes periódicas o estacionales. En particular cómo determinar sus frecuencias.
Algunos casos de regresión no lineal.
Ejemplo: relación entre mortalidad, temperatura y polución.¶
El ejemplo, tomado del libro de Shumway, consiste en tres series:
\(M_t = \) mortalidad cardiovascular semanal reportada en Los Angeles durante 10 años.
\(T_t = \) temperatura media registrada en Farenheit.
\(P_t = \) conteo de niveles de partículas en el aire.
Grafiquemos las 3 series:
M=astsa.cmort
T=astsa.tempr
P=astsa.part
fig, axs = plt.subplots(3, 1, figsize=[10,6])
M.plot(ax=axs[0], title="Mortalidad Cardiovascular", legend=False)
T.plot(ax=axs[1], title="Temperatura", color="teal", legend=False);
P.plot(ax=axs[2], xlabel="Año", title="Partículas", color="firebrick", legend=False);
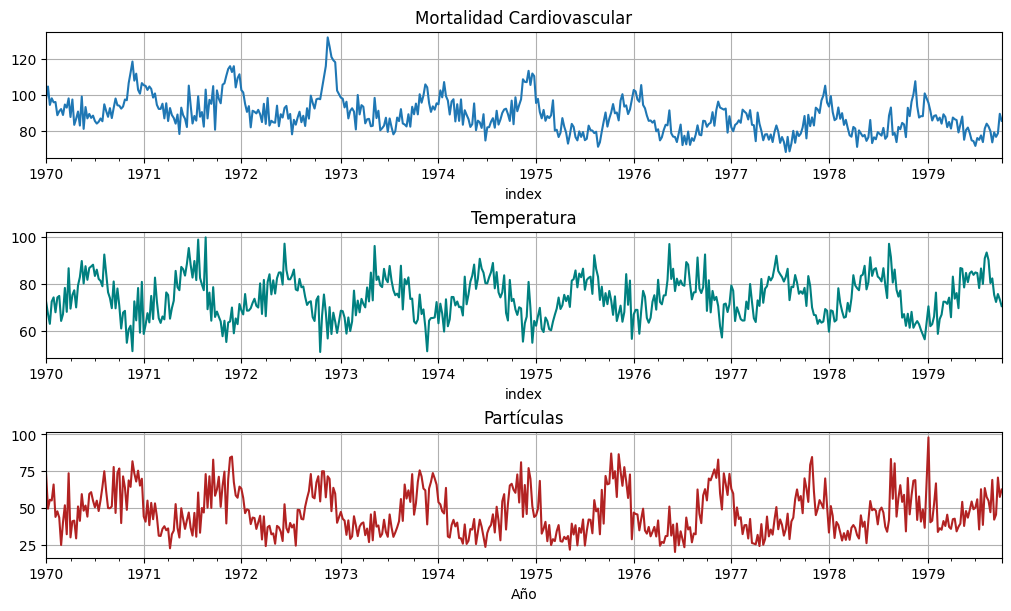
Observaciones:
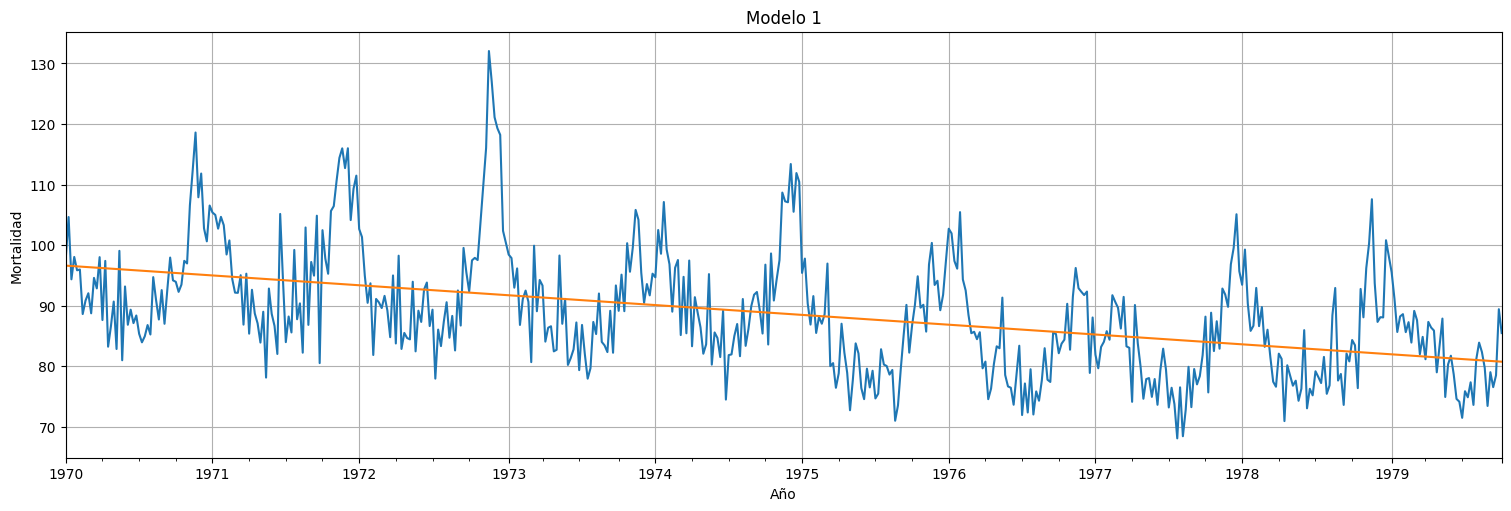
Se ve una tendencia decreciente de mortalidad.
Se ven ciclos estacionales claros (anuales)
Mortalidad y temperatura parecen estar “en contrafase” -> la gente muere más en invierno?
Temperatura y partículas parecen estar “en contrafase” también -> la lluvia en invierno limpia?
Para analizar estas relaciones se puede hacer un gráfico de correlaciones, con el comando scatter_matrix de pandas.plotting.
import pandas as pd
import numpy as np
import scipy as sp
data = pd.concat([M, T, P], axis=1)
data.columns = ["Mortalidad", "Temperatura", "Particulas"]
pd.plotting.scatter_matrix(data, figsize=(9, 6), diagonal="kde") #kde pone estimaciones de la densidad en la diagonal
plt.show()
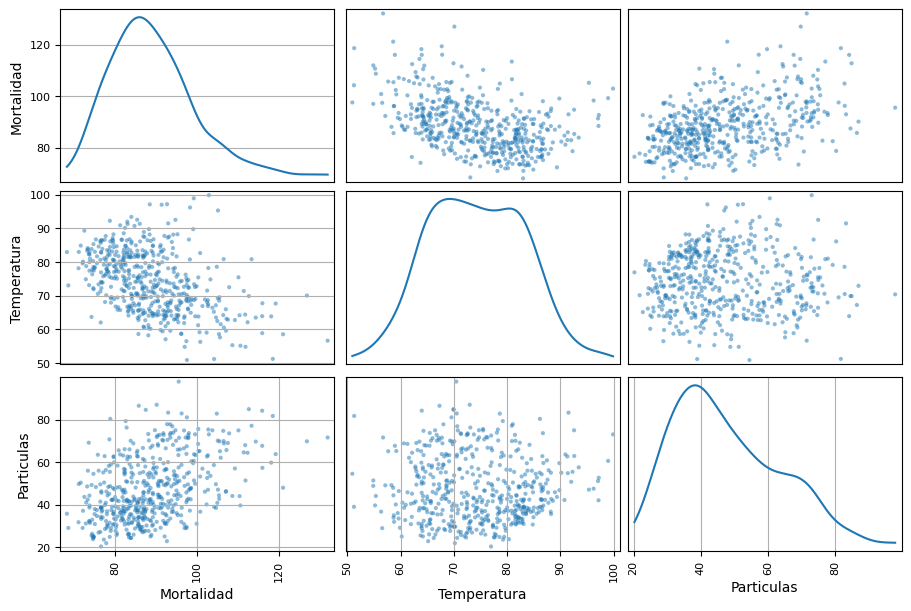
Se propone entonces ajustar algunos modelos que buscan explicar la serie de mortalidad a partir de las otras:
\(M_t = \beta_0 + \beta_1 t + w_t.\)
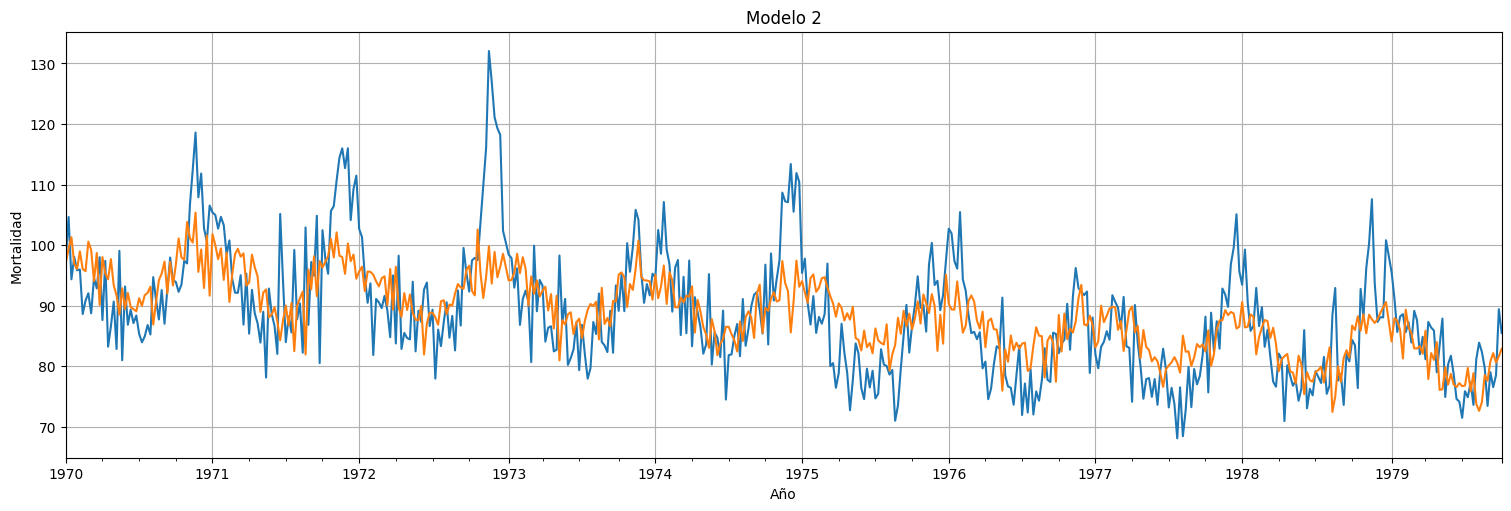
\(M_t = \beta_0 + \beta_1 t + \beta_2(T_t - \bar{T}) + w_t.\)
\(M_t = \beta_0 + \beta_1 t + \beta_2(T_t - \bar{T}) + \beta_3(T_t - \bar{T})^2 + w_t.\)
\(M_t = \beta_0 + \beta_1 t + \beta_2(T_t - \bar{T}) + \beta_3(T_t - \bar{T})^2 + \beta_4 P_t + w_t.\)
Modelo 1¶
import statsmodels.api as sm #para el qqplot
from statsmodels.formula.api import ols
time = pd.Series([idx.ordinal for idx in M.index], index=M.index, name="Semana")
datos = pd.concat([time,M], axis=1)
datos.columns = ["Semana","Mortalidad"]
fit = ols(formula="M~Semana", data=datos).fit()
fit.summary()
| Dep. Variable: | M | R-squared: | 0.211 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.209 |
| Method: | Least Squares | F-statistic: | 135.0 |
| Date: | Tue, 22 Apr 2025 | Prob (F-statistic): | 8.03e-28 |
| Time: | 15:38:03 | Log-Likelihood: | -1829.9 |
| No. Observations: | 508 | AIC: | 3664. |
| Df Residuals: | 506 | BIC: | 3672. |
| Df Model: | 1 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| Intercept | 96.6510 | 0.790 | 122.335 | 0.000 | 95.099 | 98.203 |
| Semana | -0.0312 | 0.003 | -11.618 | 0.000 | -0.036 | -0.026 |
| Omnibus: | 67.579 | Durbin-Watson: | 0.576 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 97.699 |
| Skew: | 0.906 | Prob(JB): | 6.09e-22 |
| Kurtosis: | 4.156 | Cond. No. | 590. |
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
ax = M.plot(xlabel="Año", ylabel="Mortalidad", legend=False)
ax.plot(fit.fittedvalues)
plt.title("Modelo 1")
plt.ylabel("Mortalidad")
plt.xlabel("Año");
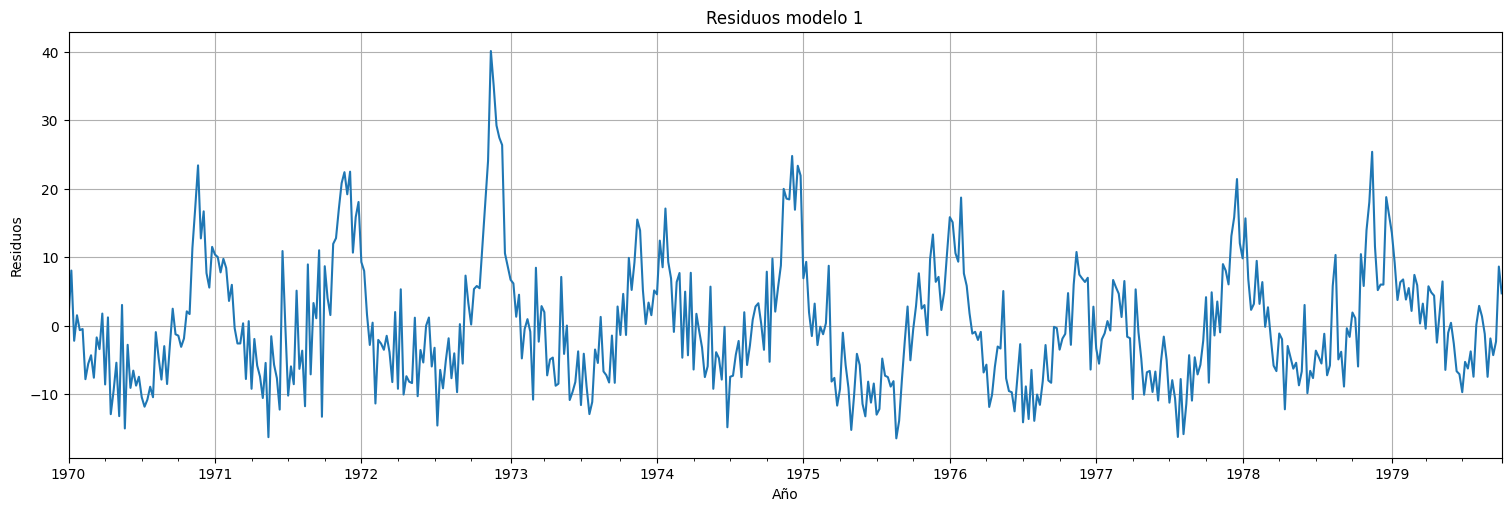
fit.resid.plot()
plt.title("Residuos modelo 1")
plt.ylabel("Residuos")
plt.xlabel("Año");
Modelo 2¶
# Modelo 2
temp = T-np.mean(T) # temperatura centrada
datos = pd.concat([time,M, temp], axis=1)
datos.columns = ["Semana","Mortalidad", "Temperatura"]
fit = ols(formula="M~Semana+Temperatura", data=datos).fit()
fit.summary()
| Dep. Variable: | M | R-squared: | 0.380 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.378 |
| Method: | Least Squares | F-statistic: | 155.0 |
| Date: | Tue, 22 Apr 2025 | Prob (F-statistic): | 3.28e-53 |
| Time: | 15:38:03 | Log-Likelihood: | -1768.4 |
| No. Observations: | 508 | AIC: | 3543. |
| Df Residuals: | 505 | BIC: | 3555. |
| Df Model: | 2 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| Intercept | 96.2251 | 0.702 | 137.154 | 0.000 | 94.847 | 97.604 |
| Semana | -0.0295 | 0.002 | -12.377 | 0.000 | -0.034 | -0.025 |
| Temperatura | -0.4579 | 0.039 | -11.763 | 0.000 | -0.534 | -0.381 |
| Omnibus: | 58.204 | Durbin-Watson: | 1.214 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 80.792 |
| Skew: | 0.818 | Prob(JB): | 2.86e-18 |
| Kurtosis: | 4.068 | Cond. No. | 591. |
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
ax = M.plot(xlabel="Año", ylabel="Mortalidad", legend=False)
ax.plot(fit.fittedvalues)
plt.title("Modelo 2")
plt.ylabel("Mortalidad")
plt.xlabel("Año");
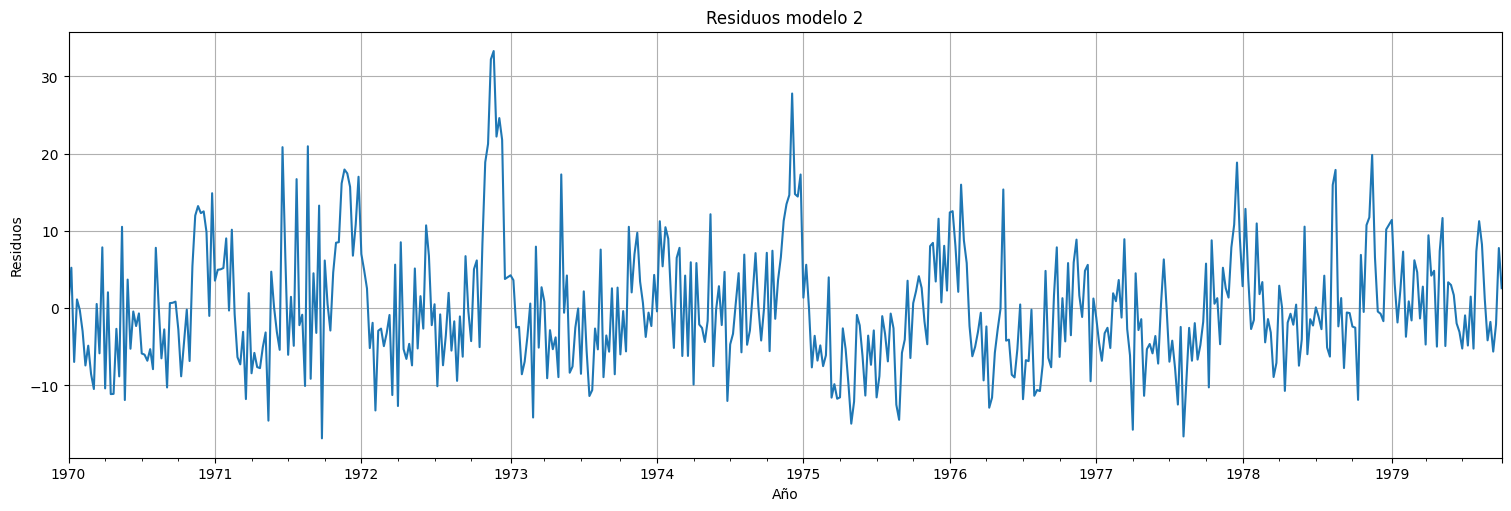
fit.resid.plot()
plt.title("Residuos modelo 2")
plt.ylabel("Residuos")
plt.xlabel("Año");
Modelo 3¶
temp2 = temp**2 # cuadrados
datos = pd.concat([time,M, temp, temp2], axis=1)
datos.columns = ["Semana","Mortalidad", "Temperatura", "Temperatura2"]
fit = ols(formula="Mortalidad~Semana+Temperatura+Temperatura2", data=datos).fit()
fit.summary()
| Dep. Variable: | Mortalidad | R-squared: | 0.448 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.445 |
| Method: | Least Squares | F-statistic: | 136.3 |
| Date: | Tue, 22 Apr 2025 | Prob (F-statistic): | 1.14e-64 |
| Time: | 15:38:03 | Log-Likelihood: | -1739.1 |
| No. Observations: | 508 | AIC: | 3486. |
| Df Residuals: | 504 | BIC: | 3503. |
| Df Model: | 3 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
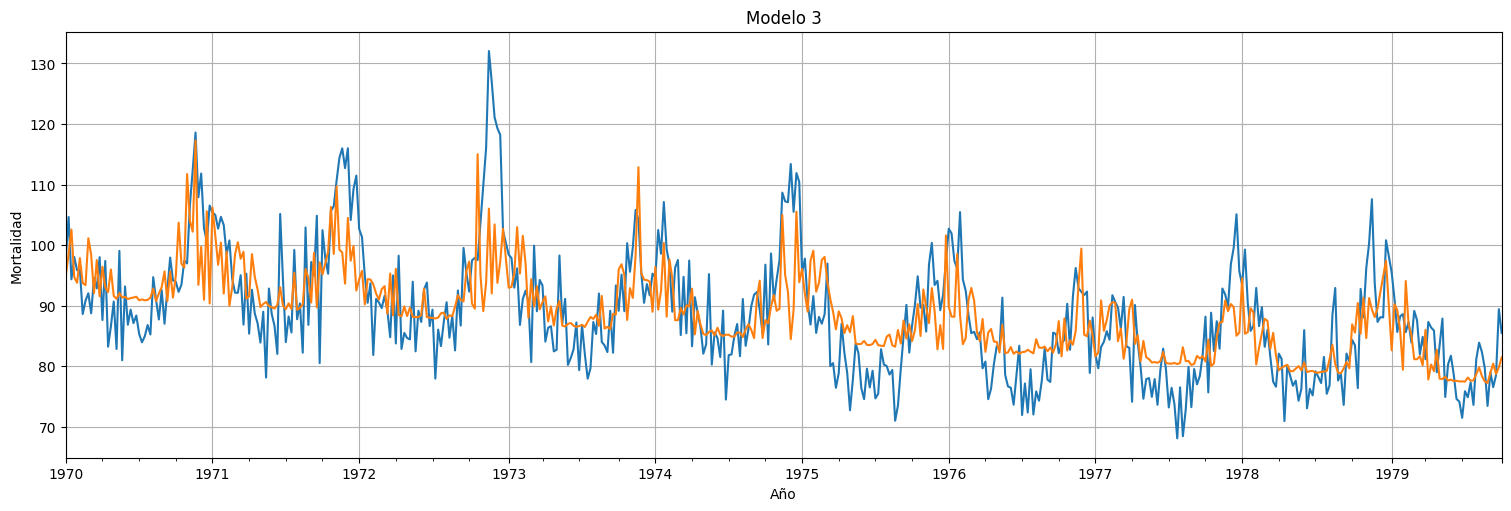
| Intercept | 93.9179 | 0.725 | 129.543 | 0.000 | 92.493 | 95.342 |
| Semana | -0.0286 | 0.002 | -12.714 | 0.000 | -0.033 | -0.024 |
| Temperatura | -0.4808 | 0.037 | -13.032 | 0.000 | -0.553 | -0.408 |
| Temperatura2 | 0.0258 | 0.003 | 7.857 | 0.000 | 0.019 | 0.032 |
| Omnibus: | 64.188 | Durbin-Watson: | 1.326 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 99.520 |
| Skew: | 0.826 | Prob(JB): | 2.45e-22 |
| Kurtosis: | 4.405 | Cond. No. | 666. |
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
ax = M.plot(xlabel="Año", ylabel="Mortalidad", legend=False)
ax.plot(fit.fittedvalues)
plt.title("Modelo 3")
plt.ylabel("Mortalidad")
plt.xlabel("Año");
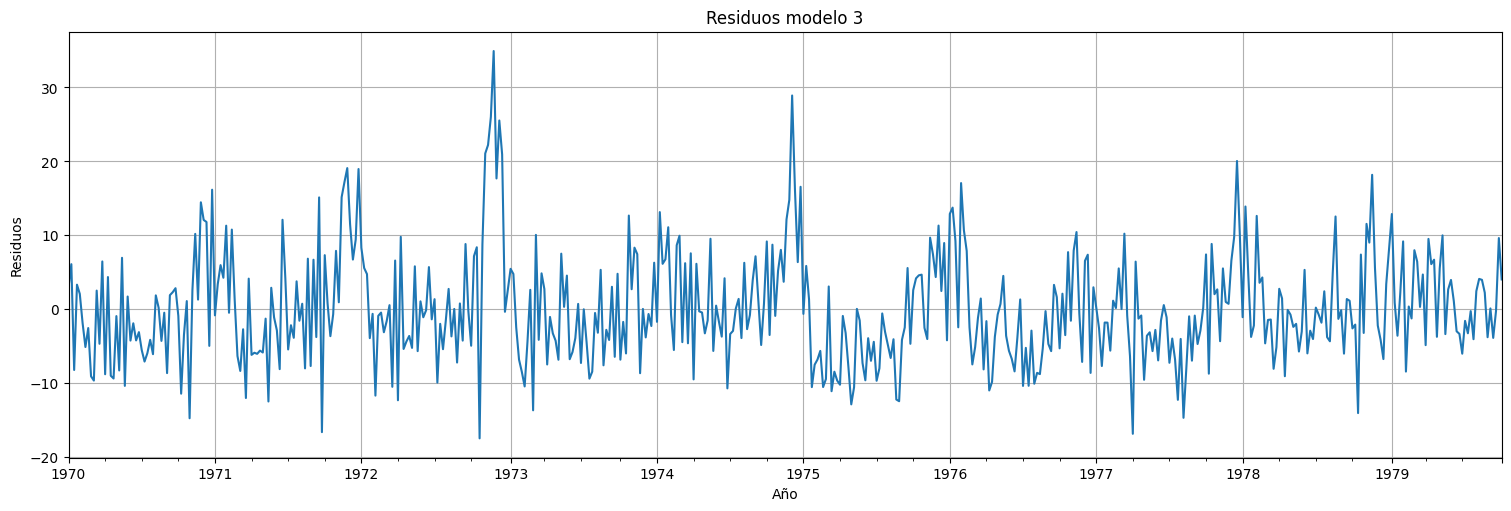
fit.resid.plot()
plt.title("Residuos modelo 3")
plt.ylabel("Residuos")
plt.xlabel("Año");
Modelo 4¶
datos = pd.concat([time,M, temp, temp2, P-np.mean(P)], axis=1)
datos.columns = ["Semana","Mortalidad", "Temperatura", "Temperatura2", "Particulas"]
fit = ols(formula="Mortalidad~Semana+Temperatura+Temperatura2+Particulas", data=datos).fit()
fit.summary()
| Dep. Variable: | Mortalidad | R-squared: | 0.595 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.592 |
| Method: | Least Squares | F-statistic: | 185.1 |
| Date: | Tue, 22 Apr 2025 | Prob (F-statistic): | 2.20e-97 |
| Time: | 15:38:04 | Log-Likelihood: | -1660.1 |
| No. Observations: | 508 | AIC: | 3330. |
| Df Residuals: | 503 | BIC: | 3351. |
| Df Model: | 4 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
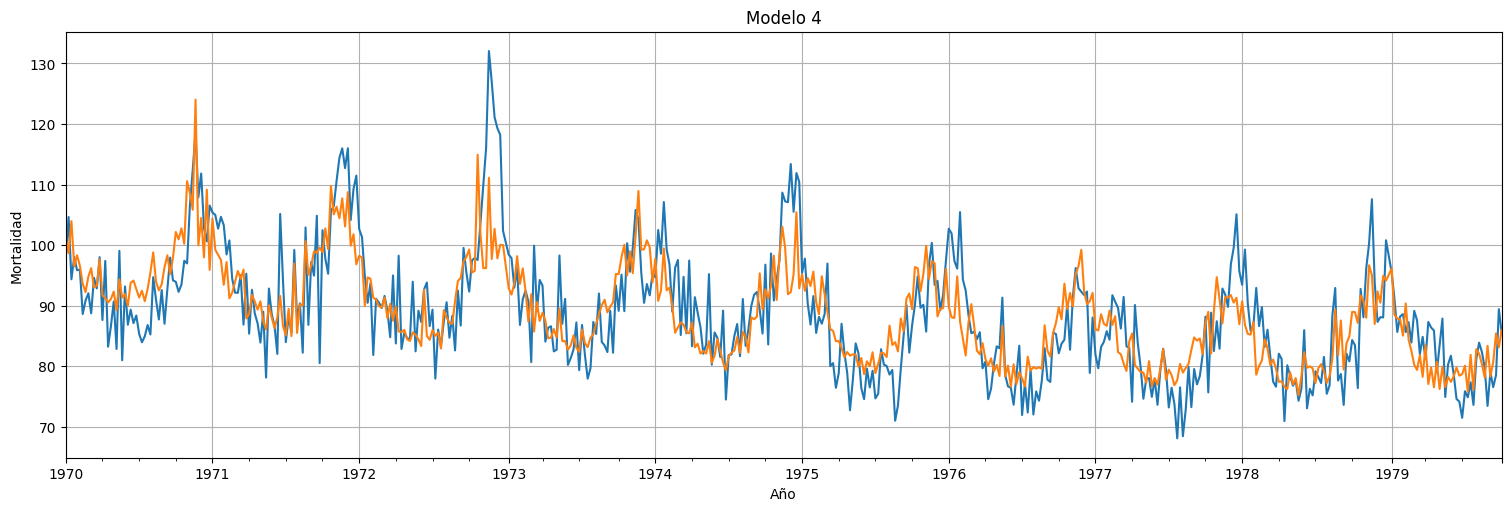
| Intercept | 93.6979 | 0.622 | 150.760 | 0.000 | 92.477 | 94.919 |
| Semana | -0.0268 | 0.002 | -13.821 | 0.000 | -0.031 | -0.023 |
| Temperatura | -0.4725 | 0.032 | -14.941 | 0.000 | -0.535 | -0.410 |
| Temperatura2 | 0.0226 | 0.003 | 7.989 | 0.000 | 0.017 | 0.028 |
| Particulas | 0.2553 | 0.019 | 13.539 | 0.000 | 0.218 | 0.292 |
| Omnibus: | 47.363 | Durbin-Watson: | 1.311 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 69.807 |
| Skew: | 0.661 | Prob(JB): | 6.94e-16 |
| Kurtosis: | 4.245 | Cond. No. | 666. |
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
ax = M.plot(xlabel="Año", ylabel="Mortalidad", legend=False)
ax.plot(fit.fittedvalues)
plt.title("Modelo 4")
plt.ylabel("Mortalidad")
plt.xlabel("Año");
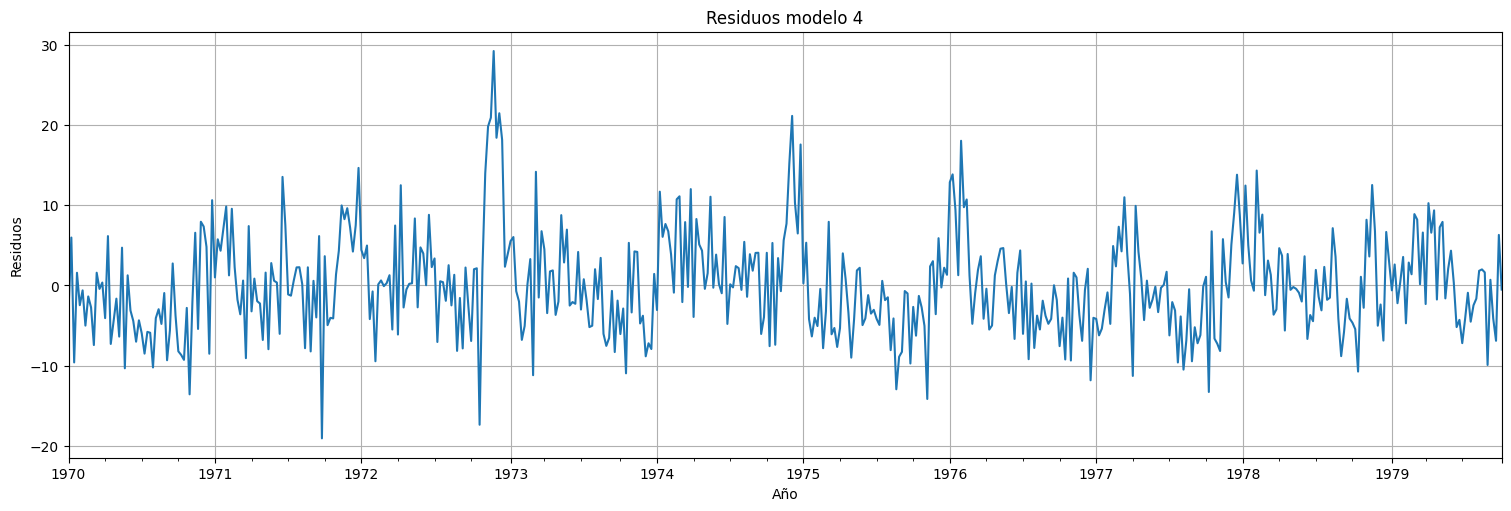
fit.resid.plot()
plt.title("Residuos modelo 4")
plt.ylabel("Residuos")
plt.xlabel("Año");
## QQ-plot es una verificación de gaussianidad.
sm.qqplot(fit.resid, line="s");
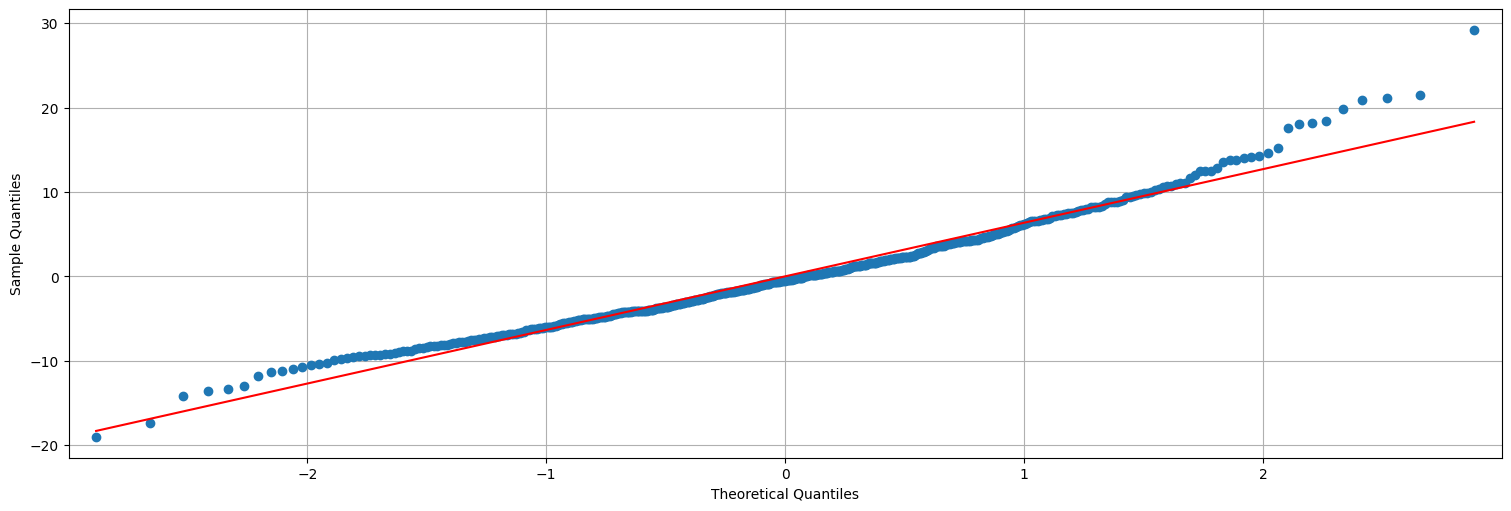
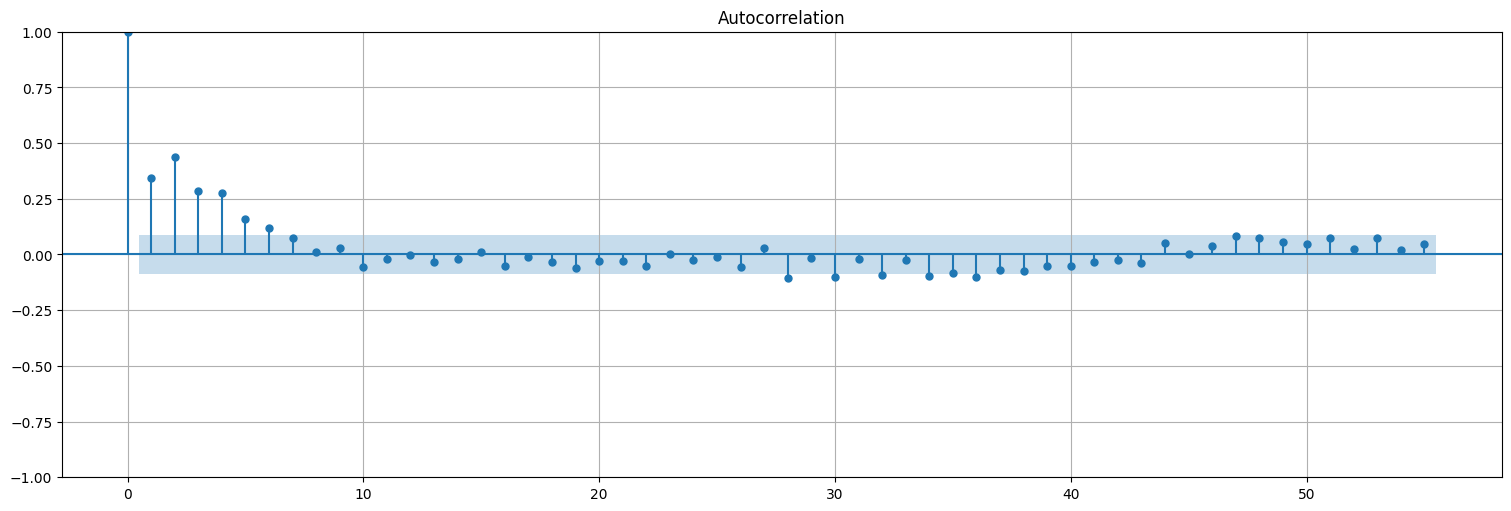
Nota:
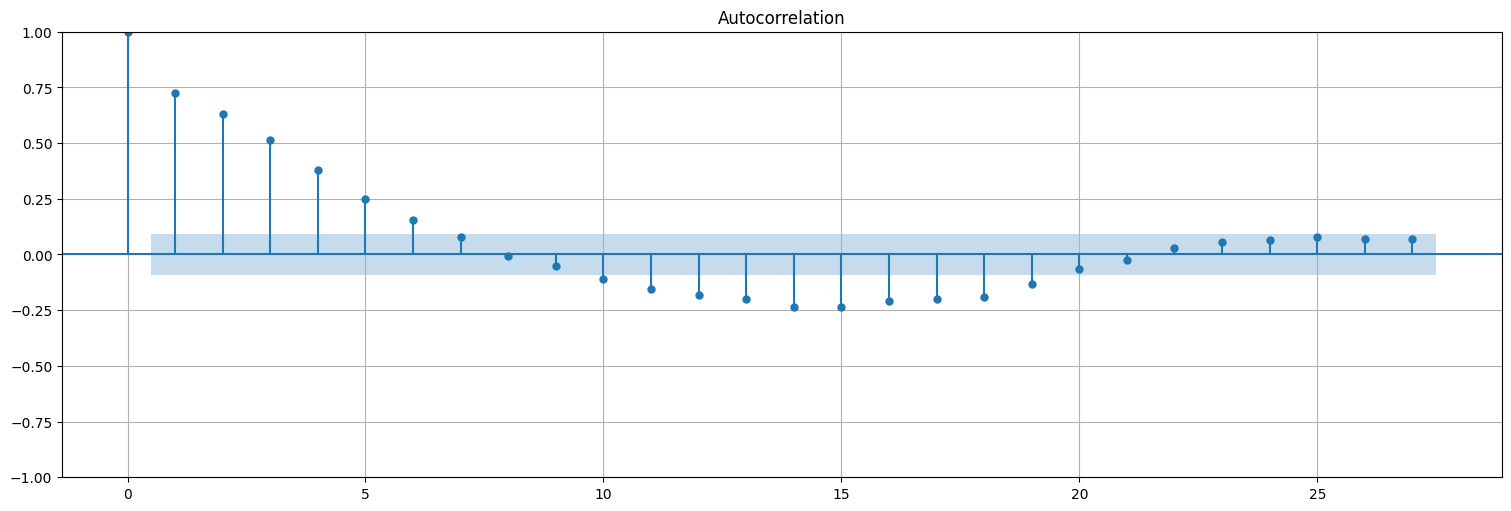
Para calcular la acf, conviene utilizar
statsmodels.tsa.graphics.plot_acf()con la opciónbartlett_confint=Falsepara usar el intervalo de confianza correcto
from statsmodels.graphics.tsaplots import plot_acf
plot_acf(fit.resid, bartlett_confint=False, lags=55);
Agreguemos ahora un modelo donde usamos también la variable \(P_t^2\) además de \(P_t\), para ver cómo el modelo comienza a empeorar en términos del \(AIC/BIC\).
# Modelo 5 (para ver que el AIC/BIC empeora)
P2 = P**2 # particulas al cuadrado
datos = pd.concat([time,M, temp, temp2, P, P2], axis=1)
datos.columns = ["Semana","Mortalidad", "Temperatura", "Temperatura2", "Particulas", "Particulas2"]
fit = ols(formula="M~Semana+Temperatura+Temperatura2+Particulas+Particulas2", data=datos).fit()
fit.summary()
| Dep. Variable: | M | R-squared: | 0.596 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.592 |
| Method: | Least Squares | F-statistic: | 148.2 |
| Date: | Tue, 22 Apr 2025 | Prob (F-statistic): | 2.06e-96 |
| Time: | 15:38:04 | Log-Likelihood: | -1659.7 |
| No. Observations: | 508 | AIC: | 3331. |
| Df Residuals: | 502 | BIC: | 3357. |
| Df Model: | 5 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| Intercept | 78.9871 | 2.987 | 26.445 | 0.000 | 73.119 | 84.855 |
| Semana | -0.0267 | 0.002 | -13.801 | 0.000 | -0.031 | -0.023 |
| Temperatura | -0.4756 | 0.032 | -14.955 | 0.000 | -0.538 | -0.413 |
| Temperatura2 | 0.0221 | 0.003 | 7.726 | 0.000 | 0.017 | 0.028 |
| Particulas | 0.3672 | 0.121 | 3.042 | 0.002 | 0.130 | 0.604 |
| Particulas2 | -0.0011 | 0.001 | -0.939 | 0.348 | -0.003 | 0.001 |
| Omnibus: | 46.388 | Durbin-Watson: | 1.309 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 68.681 |
| Skew: | 0.648 | Prob(JB): | 1.22e-15 |
| Kurtosis: | 4.251 | Cond. No. | 3.11e+04 |
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
[2] The condition number is large, 3.11e+04. This might indicate that there are
strong multicollinearity or other numerical problems.
Ejemplo: Regresión con “lag”.¶
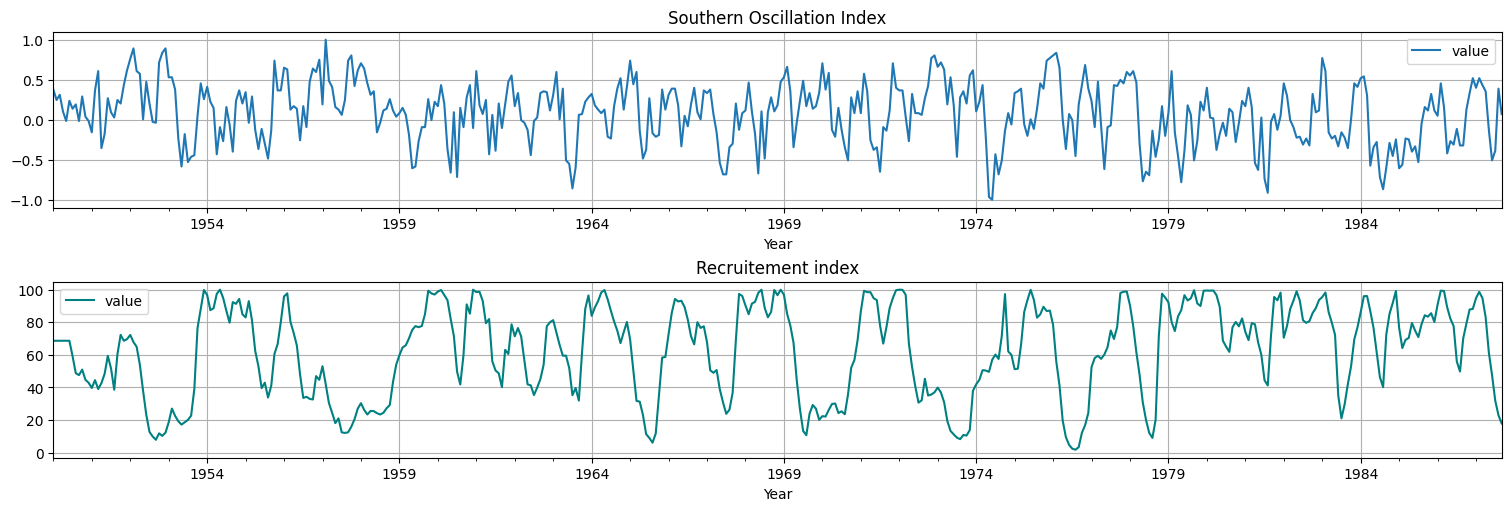
En clases anteriores vimos (a ojo) que existe una relación entre el índice del niño (Southern oscillation index, SOI) y la población de peces (Recruitement).
Recordemos las series:
soi = astsa.soi
rec = astsa.rec
fig, axs = plt.subplots(2, 1, constrained_layout = True)
soi.plot(ax=axs[0], xlabel="Year", title="Southern Oscillation Index")
rec.plot(ax=axs[1], xlabel="Year", title="Recruitement index", color="teal");
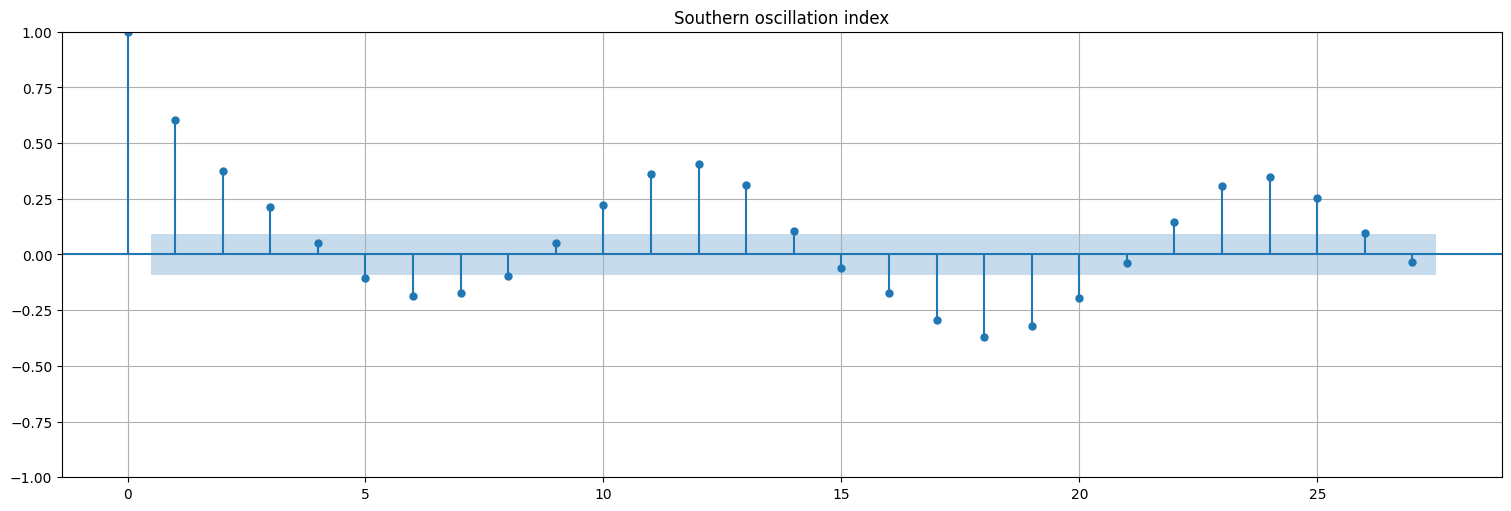
Analicemos la autocorrelación de cada serie, y la correlación cruzada:
plot_acf(soi, bartlett_confint=False);
plt.title("Southern oscillation index");
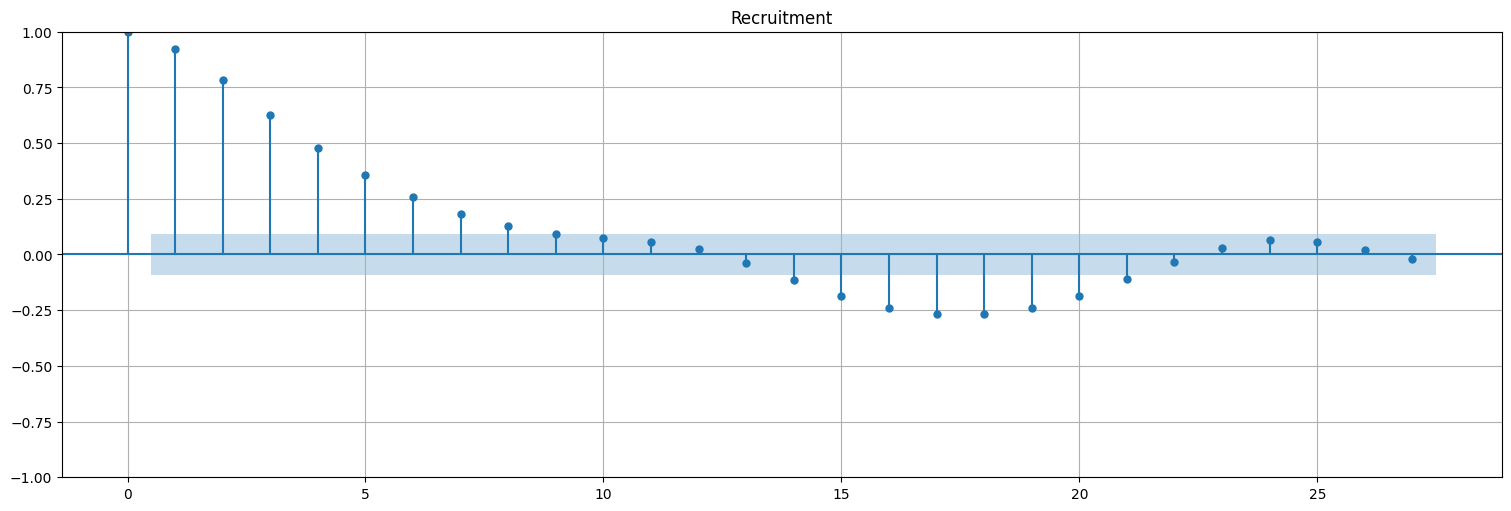
plot_acf(rec, bartlett_confint=False)
plt.title("Recruitment");
Cálculo de correlación cruzada :¶
Esta función grafica la correlación cruzada de manera adecuada, considerando lags positivos y negativos,
def ccf(x, y, max_lag, ax=None, **kwargs):
lags = np.arange(-max_lag, max_lag + 1)
backwards = sm.tsa.ccf(x[::-1], y[::-1], adjusted=False)[max_lag::-1]
forwards = sm.tsa.ccf(x, y, adjusted=False)[:max_lag + 1]
ccf = np.r_[backwards[:-1], forwards]
ylabel = "CCF"
if ax is None:
ax = plt.gca()
ax.stem(lags, ccf, basefmt="k")
ax.axvline(0, color="black", linestyle="--", linewidth=1)
conf_level = 1.96 / np.sqrt(x.shape[0])
ax.fill_between(lags,conf_level,-conf_level,alpha=0.25)
ax.set_xlabel("LAG")
ax.set_ylabel(ylabel)
return ax
ccf(soi,rec, max_lag=30)
plt.title("SOI vs Recruitment");
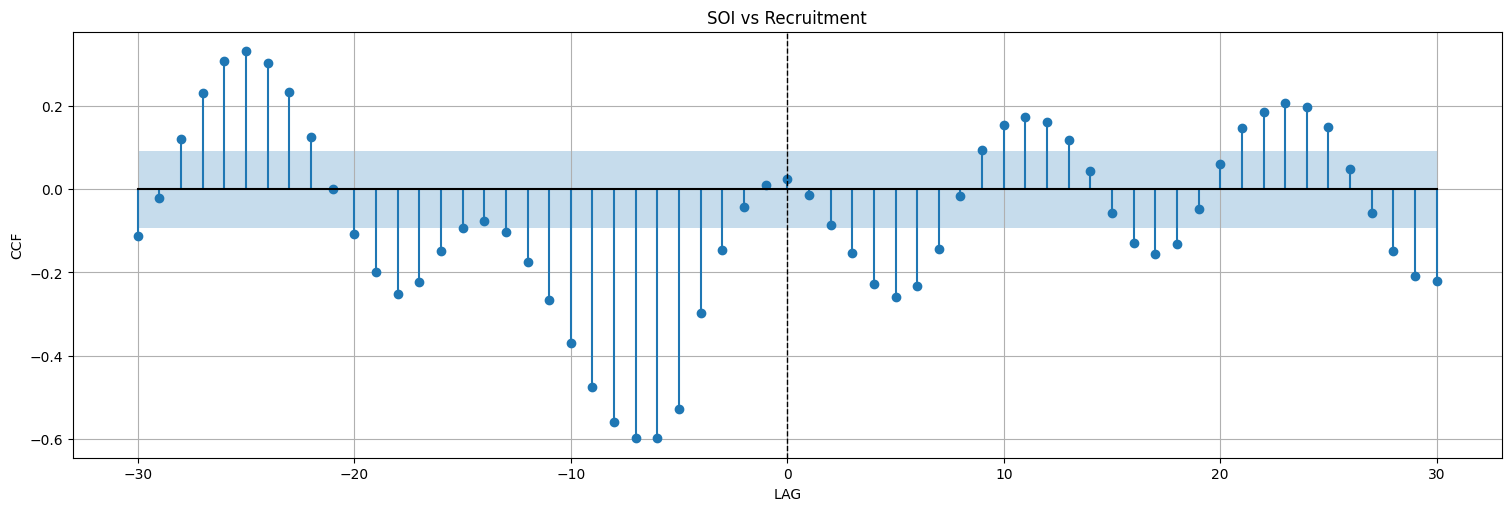
Observaciones:
La serie del SOI muestra correlaciones fuertes cada 12 meses.
El libro dice que la de recruitement también pero yo no las veo (más adelante retomamos esto).
La correlación cruzada tiene un pico (negativo) en -6 meses. Esto indica que la serie SOI puede predecir la serie de Recruitement usando el valor de 6 meses antes.
Se propone entonces un modelo de la forma:
Es decir, estimar el recruitement actual por una media más algo que depende del SOI \(6\) meses antes. Observemos que esto es un modelo lineal como antes, donde simplmente figuran como “features” el intercept u ordenada en el origen, y los valores de \(S\) corridos.
Problema: ¿como logramos alinear la serie \(R_t\) en tiempo \(t\) y la serie \(S_{t-6}\) laggeada?
Usamos el comando
shiftpara correr la serie
lag=6
plt.scatter(soi.shift(lag), rec, label="datos");
#Agrego un suavizado local para ver la tendencia
lowess = sm.nonparametric.lowess(rec.iloc[:, 0], soi.iloc[:, 0].shift(lag))
plt.plot(lowess[:, 0], lowess[:, 1], color="firebrick", label="loess");
plt.legend()
plt.xlabel("SOI(t-6)")
plt.ylabel("Rec(t)");
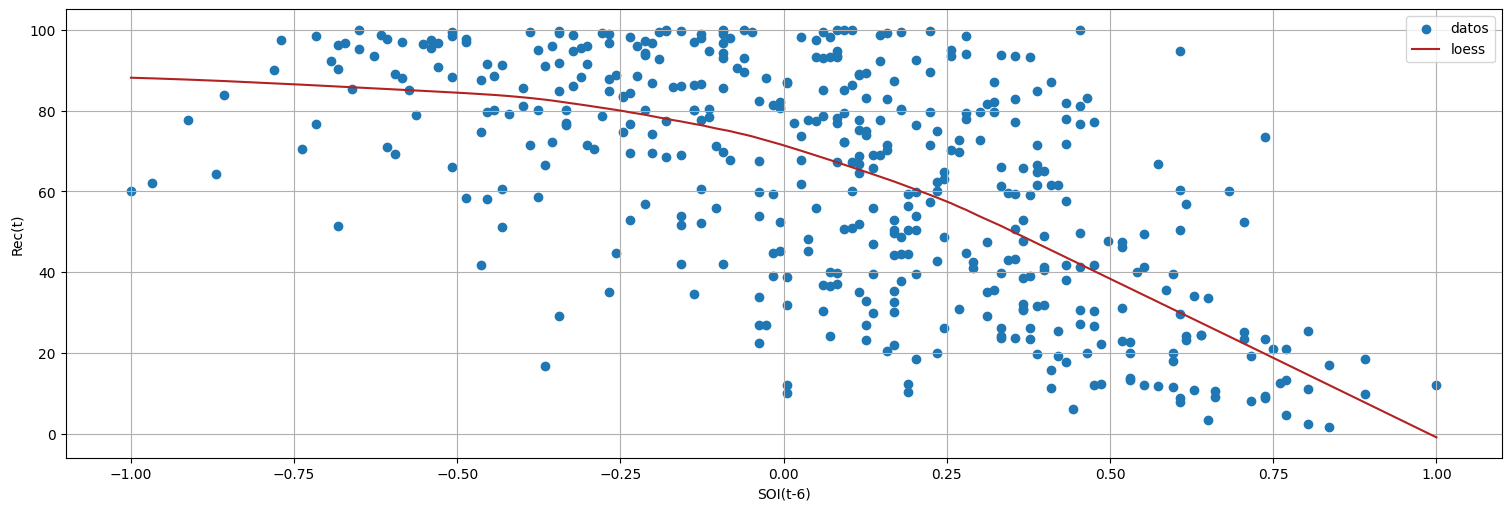
Nota: la curva roja es una regresión “loess” simple para ver aproximadamente la corelación.
#Podemos llamar a fit haciendo referencia a las columnas del dataframe (fish en este caso).
fish = pd.concat([rec, soi.shift(6)], axis=1).dropna()
fish.columns = ["rec", "soiL6"]
fit = ols("rec ~ soiL6", data=fish).fit()
fit.summary()
| Dep. Variable: | rec | R-squared: | 0.363 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.362 |
| Method: | Least Squares | F-statistic: | 253.5 |
| Date: | Tue, 22 Apr 2025 | Prob (F-statistic): | 1.68e-45 |
| Time: | 15:38:05 | Log-Likelihood: | -2024.9 |
| No. Observations: | 447 | AIC: | 4054. |
| Df Residuals: | 445 | BIC: | 4062. |
| Df Model: | 1 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| Intercept | 65.7898 | 1.088 | 60.469 | 0.000 | 63.652 | 67.928 |
| soiL6 | -44.2826 | 2.781 | -15.923 | 0.000 | -49.748 | -38.817 |
| Omnibus: | 16.029 | Durbin-Watson: | 0.547 |
|---|---|---|---|
| Prob(Omnibus): | 0.000 | Jarque-Bera (JB): | 8.017 |
| Skew: | -0.088 | Prob(JB): | 0.0182 |
| Kurtosis: | 2.368 | Cond. No. | 2.63 |
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
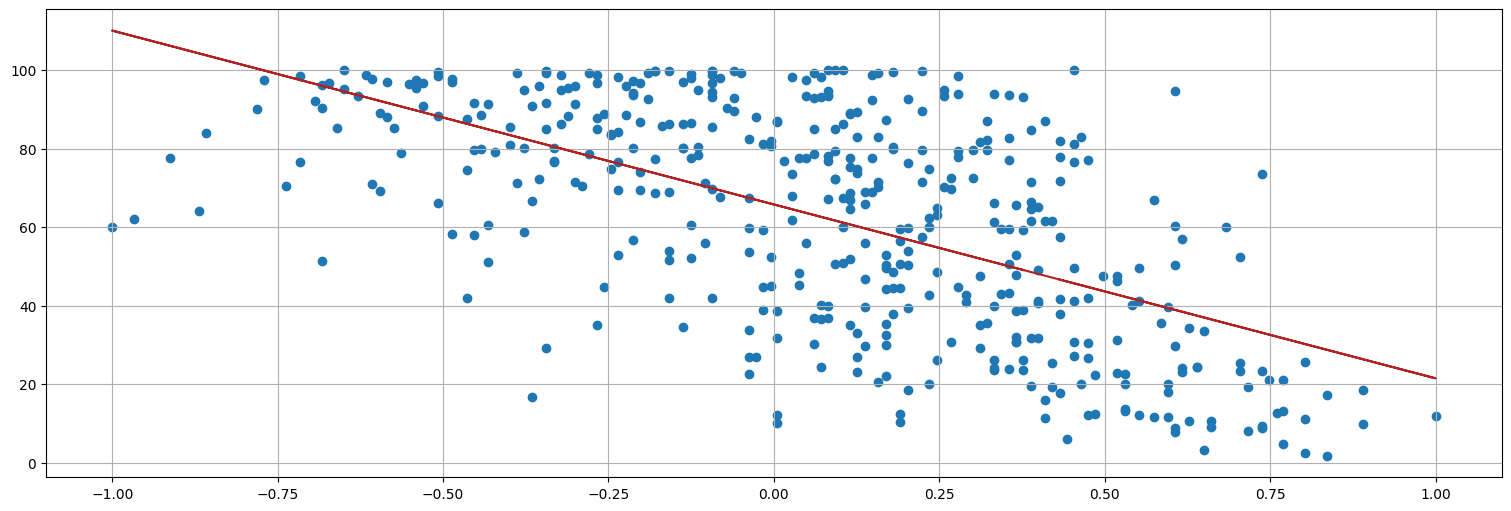
plt.plot(fish.soiL6,fit.fittedvalues, color="firebrick")
plt.scatter(soi.shift(lag), rec);
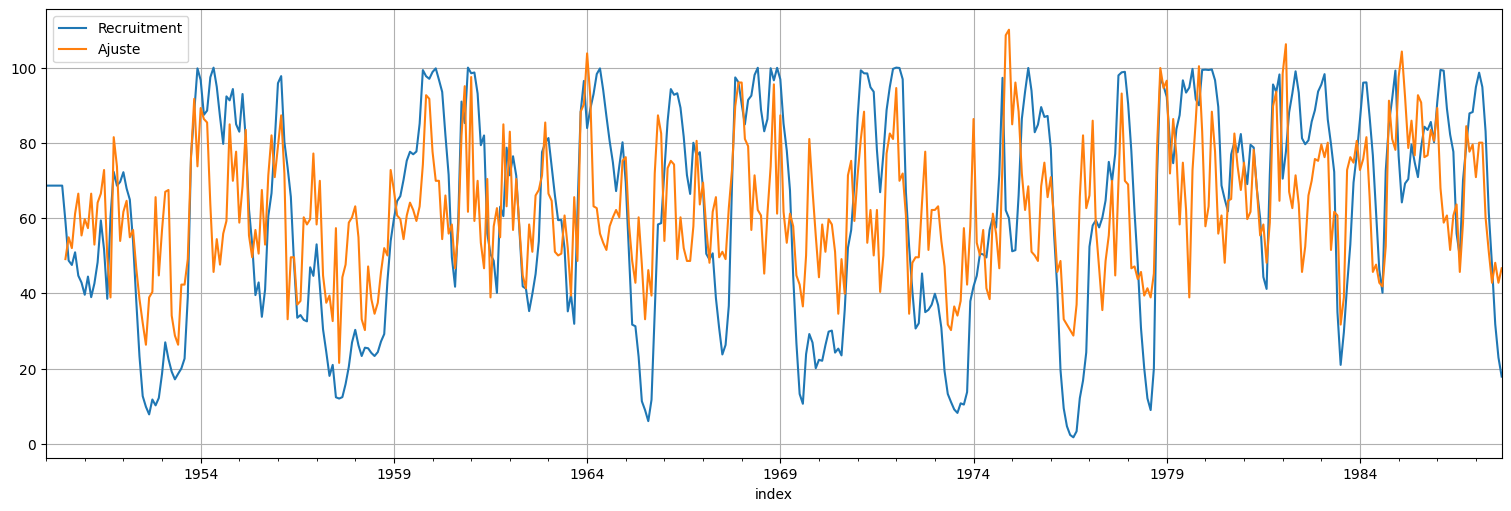
rec.plot()
fit.fittedvalues.plot().legend(["Recruitment","Ajuste"]);
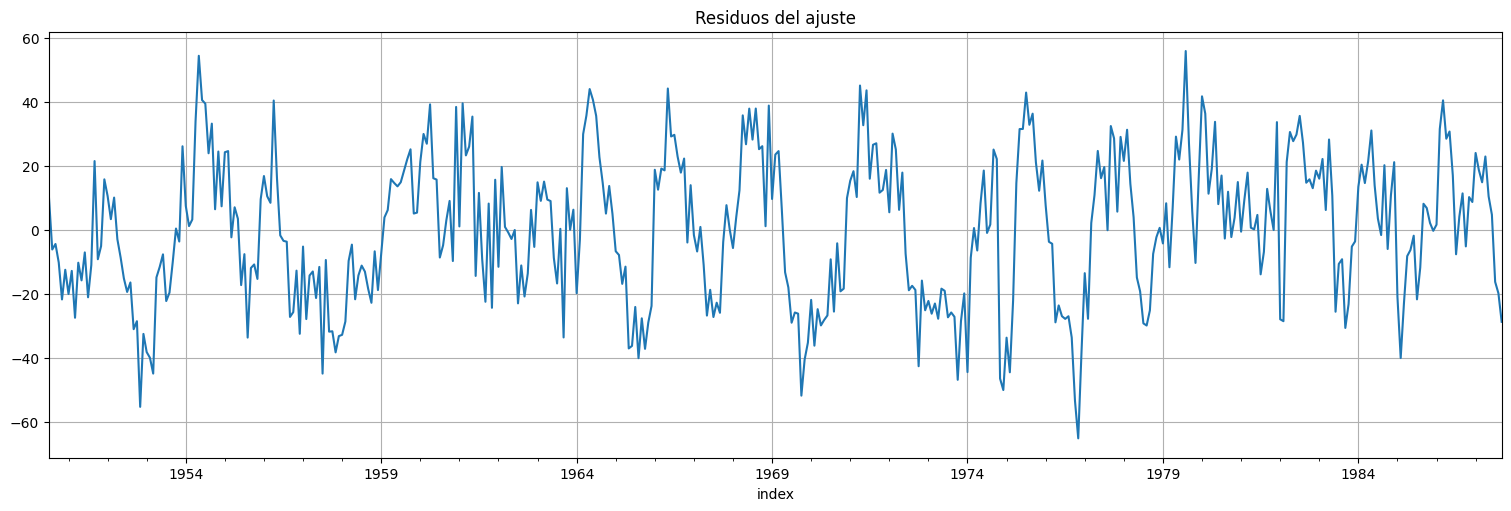
Análisis de residuos del ajuste¶
fit.resid.plot();
plt.title("Residuos del ajuste");
print(f"RMSE del residuo: {np.std(fit.resid)}")
RMSE del residuo: 22.44678221194104
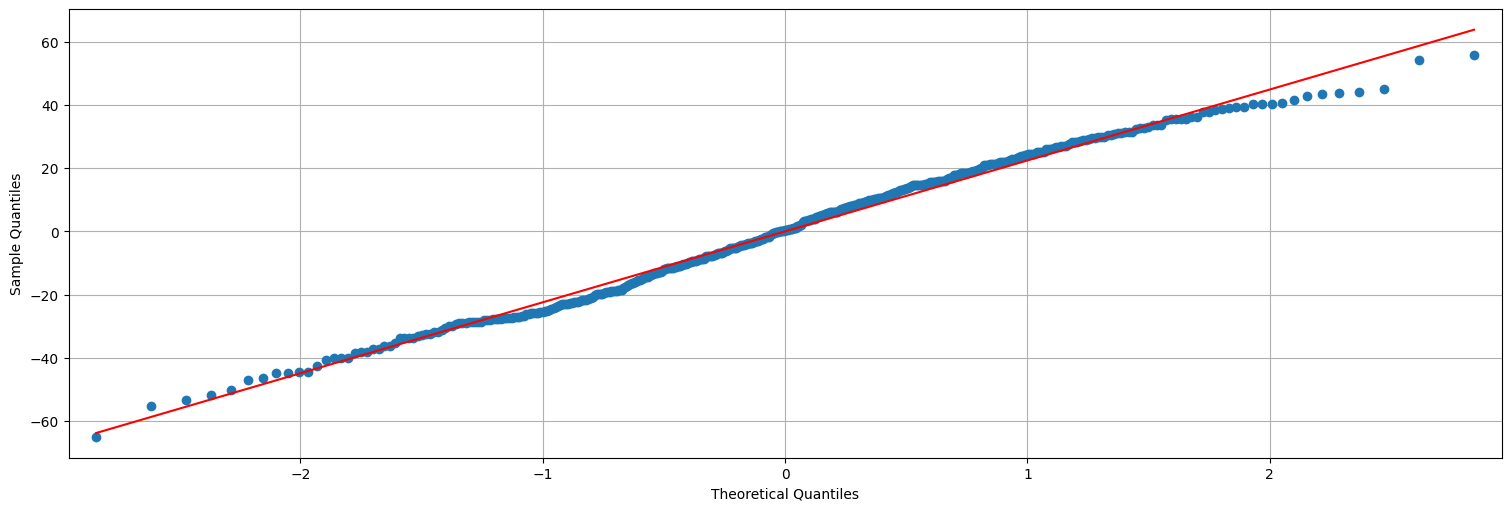
## QQ-plot es una verificación de gaussianidad.
sm.qqplot(fit.resid, line="s");
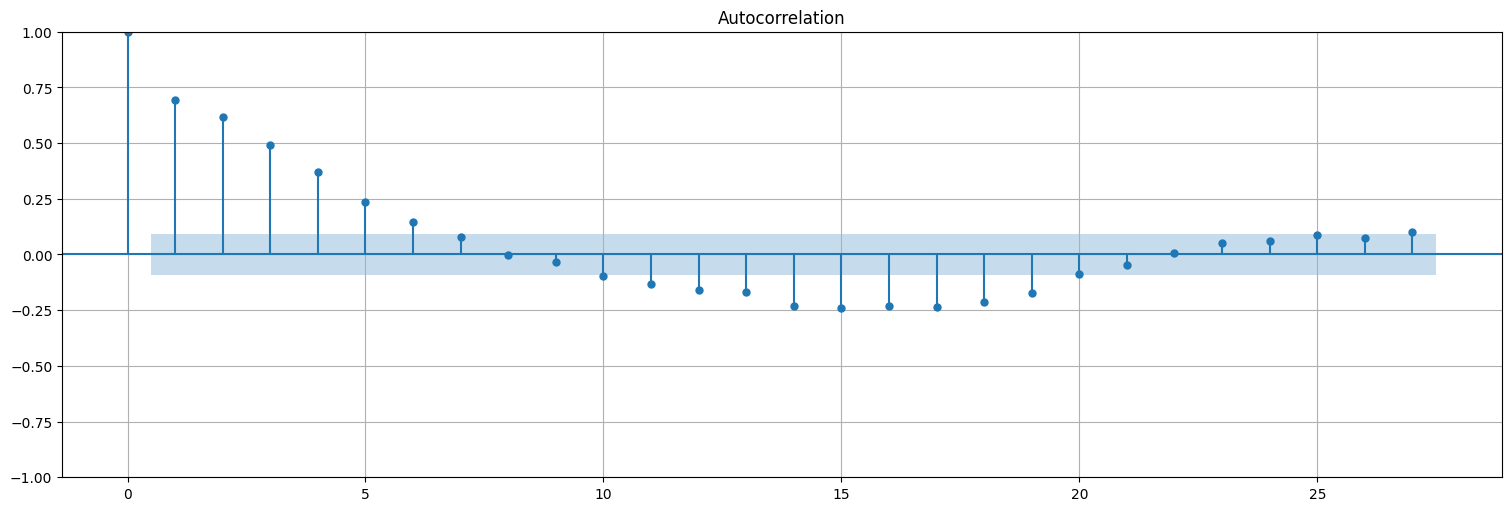
plot_acf(fit.resid, bartlett_confint=False);
Mejorando el modelo¶
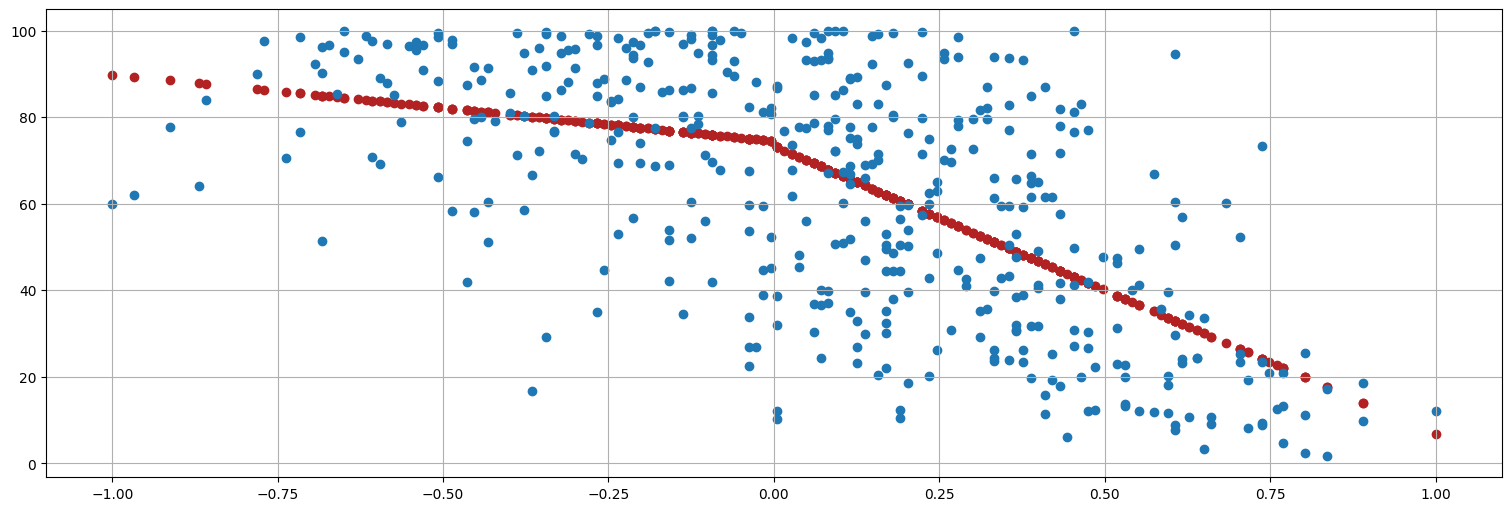
De la gráfica de \(R_t\) vs. \(S_{t-6}\) queda claro que el comportamiento es distinto dependiendo de si es positivo o negativo el valor de SOI. Podemos mejorar el modelo incluyendo una variable indicatriz para esto, es decir:
siendo \(D_t = 1\) si \(S_t>0\) y \(0\) en otro caso. Esto es equivalente a ajustar dos rectas en las dos regiones.
dummy = soi>0
fish = pd.concat([rec, soi.shift(6), dummy.shift(6)], axis=1).dropna()
fish.columns = ["rec", "soiL6", "DL6"]
fit = ols("rec ~ soiL6+DL6+soiL6*DL6", data=fish).fit()
print(f"RMSE de los residuos: {np.sqrt(fit.mse_resid)}")
fit.summary()
RMSE de los residuos: 21.83859202064757
| Dep. Variable: | rec | R-squared: | 0.402 |
|---|---|---|---|
| Model: | OLS | Adj. R-squared: | 0.398 |
| Method: | Least Squares | F-statistic: | 99.43 |
| Date: | Tue, 22 Apr 2025 | Prob (F-statistic): | 3.20e-49 |
| Time: | 15:38:06 | Log-Likelihood: | -2010.7 |
| No. Observations: | 447 | AIC: | 4029. |
| Df Residuals: | 443 | BIC: | 4046. |
| Df Model: | 3 | ||
| Covariance Type: | nonrobust |
| coef | std err | t | P>|t| | [0.025 | 0.975] | |
|---|---|---|---|---|---|---|
| Intercept | 74.4794 | 2.865 | 25.998 | 0.000 | 68.849 | 80.110 |
| DL6[T.True] | -1.1394 | 3.711 | -0.307 | 0.759 | -8.433 | 6.155 |
| soiL6 | -15.3583 | 7.401 | -2.075 | 0.039 | -29.904 | -0.812 |
| soiL6:DL6[T.True] | -51.2441 | 9.523 | -5.381 | 0.000 | -69.959 | -32.529 |
| Omnibus: | 4.767 | Durbin-Watson: | 0.610 |
|---|---|---|---|
| Prob(Omnibus): | 0.092 | Jarque-Bera (JB): | 4.682 |
| Skew: | -0.215 | Prob(JB): | 0.0962 |
| Kurtosis: | 2.741 | Cond. No. | 14.1 |
Notes:
[1] Standard Errors assume that the covariance matrix of the errors is correctly specified.
plt.scatter(fish.soiL6,fit.fittedvalues, color="firebrick")
plt.scatter(soi.shift(lag), rec);
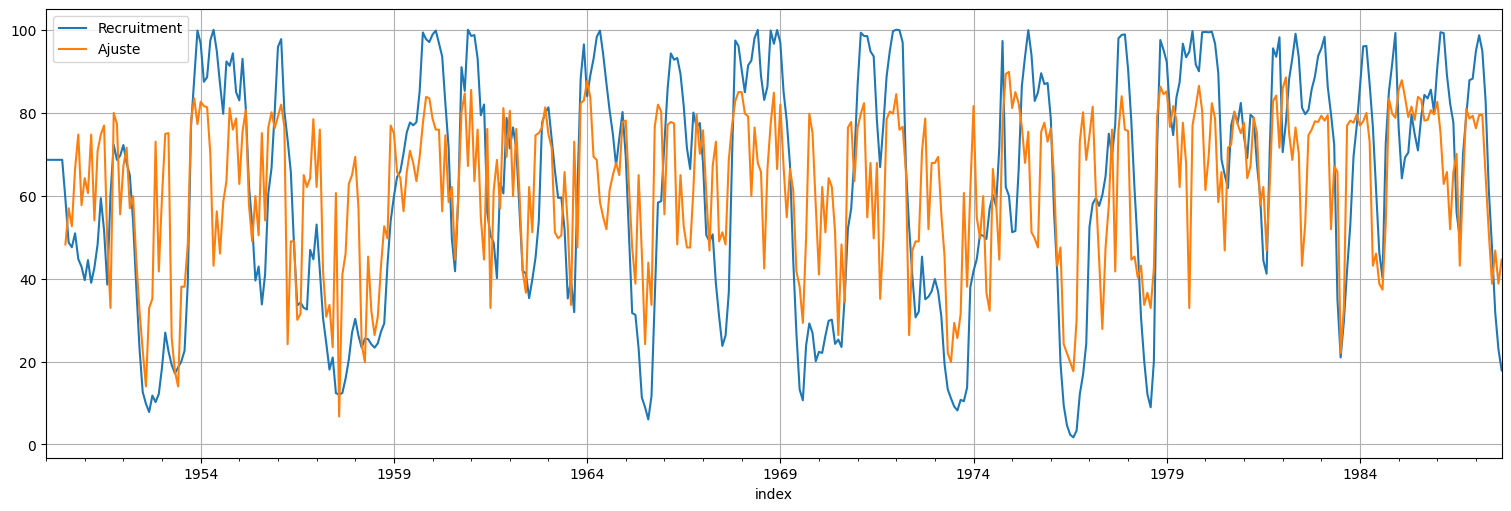
rec.plot()
fit.fittedvalues.plot().legend(["Recruitment","Ajuste"]);
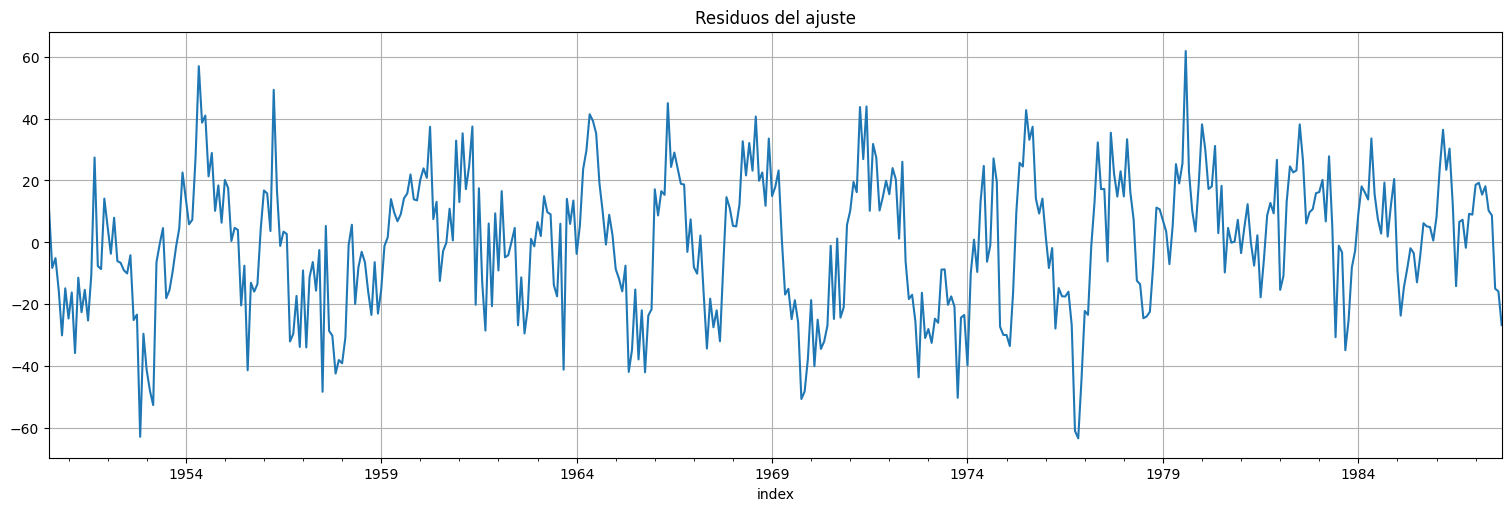
Análisis de residuos del ajuste¶
fit.resid.plot();
plt.title("Residuos del ajuste");
print(f"RMSE del residuo: {np.std(fit.resid)}")
RMSE del residuo: 21.74066062129114
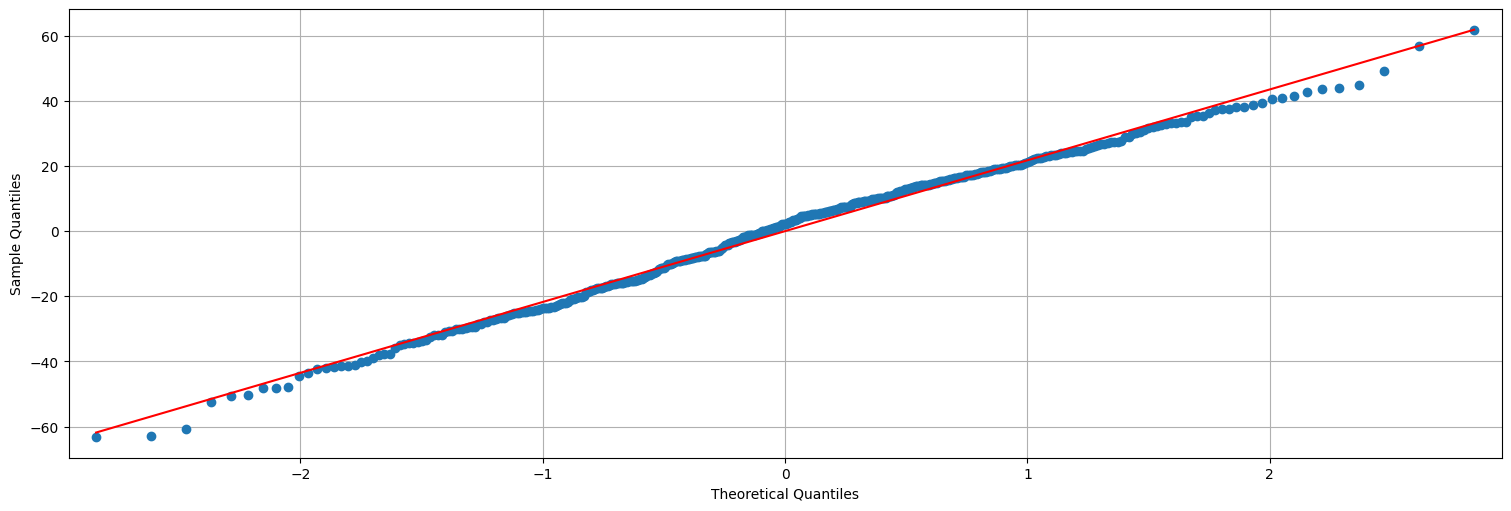
## QQ-plot es una verificación de gaussianidad.
sm.qqplot(fit.resid, line="s");
plot_acf(fit.resid, bartlett_confint=False);
Ejercicios¶
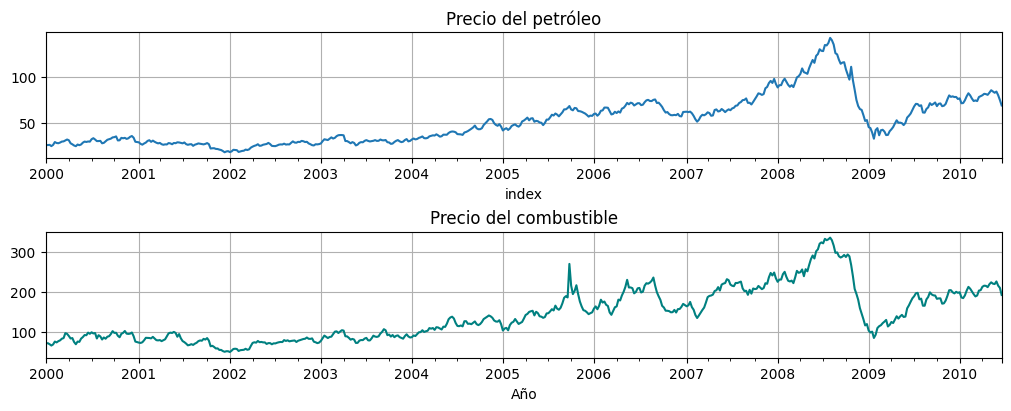
Relación entre el precio del petróleo y la gasolina.¶
Considere las dos series semanales oil (precio del petróleo) and gas (precio del combustible). La primera está en dólares por barril, la segunda en centavos por galón.
Graficar ambas en una misma gráfica. ¿Cuál de los modelos ya vistos se asemeja más? ¿Son estacionarias?
En economía, muchas veces la información se encuentra en el porcentaje de cambio de la serie (retorno o tasa de crecimiento). Argumente que la transformación \(y_t= \log(x_t) - \log(x_{t-1})\) puede usarse para obtener el retorno.
Aplicar la transformación a las series anteriores, volver a graficar y analizar la autocorrelación de las mismas.
Graficar la correlación cruzada de las series transformadas. Los valores pequeños pero significativos donde gas adelanta a oil pueden ser considerados “realimentación” entre ellas.
Realizar nubes de puntos para las series laggeadas, con hasta 3 semanas de diferencia.
Hay estudios que afirman que los precios de combustible responden más rápido a las subidas del petróleo que a las bajadas. Para explorar esto hagamos una regresión con lags simple.
Sean \(G_t\) y \(O_t\) las series transformadas de gas y oil respectivamente. Ajuste una regresión:
siendo \(I_t = 1\) si \(O_t \geqslant 0\) y \(0\) si no (\(I_t\) representa si oil está creciendo). Sugerencia: construir \(I\) como en el ejemplo de soi visto en clase.
Analizar y discutir los resultados.
oil=astsa.oil
gas=astsa.gas
fig, axs = plt.subplots(2, 1, figsize=[10,4])
oil.plot(ax=axs[0], title="Precio del petróleo", legend=False)
gas.plot(ax=axs[1], title="Precio del combustible", color="teal", xlabel="Año", legend=False);